| |
| Med Sci (Paris). 2002 April; 18(4): 429–437. Published online 2002 April 15. doi: 10.1051/medsci/2002184429.Les récepteurs nucléaires CAR et PXR contrôlent l’induction des cytochromes P450 par le phénobarbital Catherine Corcos,1 Juliette Brey,2 and Laurent Corcos2* 1Laboratoire de Biologie Moléculaire, Faculté des Sciences Gabriel, 6, boulevard Gabriel, 21000 Dijon, France 2Inserm U.517, Faculté de Médecine, 7, boulevard Jeanne d’Arc, 21000 Dijon, France |
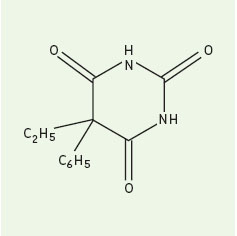
Le phénobarbital, un composé utilisé en thérapeutique anti-épileptique depuis de nombreuses années, est connu pour provoquer toute une série d’effets dans l’organisme, en particulier au niveau hépatique chez les mammifères [1, 2]. Le phénobarbital, dont la structure est schématisée sur la Figure 1, est un représentant d’une très large famille de molécules sans parenté structurale manifeste, mais fortement lipophiles. La réponse au phénobarbital se caractérise par une augmentation du poids du foie, une prolifération du réticulum endoplasmique et l’induction (ou l’inhibition) d’un grand nombre de gènes [3, 4]. Avec l’important recul de son utilisation en médecine, le phénobarbital ne semble pas impliqué dans l’apparition d’effets secondaires à la chronicité des traitements, comme l’induction de cancers [5], bien qu’il soit un promoteur de tumorigenèse chez le rat [6]. Les gènes dont l’expression est contrôlée par le phénobarbital sont pour la plupart impliqués dans la détoxification, un ensemble de voies métaboliques permettant une assimilation de molécules potentiellement toxiques telles que des médicaments ou des polluants [2, 7]. Cependant, les gènes jouant un rôle probable dans la cancérogenèse dépendante du phénobarbital codent pour des récepteurs de certains facteurs de croissance comme l’EGF (epidermal growth factor) ou des protéines des jonctions intercellulaires comme les connexines [8, 9]. Les cytochromes P450 (CYP) de la sous-famille 2B sont les enzymes sur lesquelles l’activité inductrice du phénobarbital a été initialement observée, et est la plus marquée. À l’instar d’autres xénobiotiques, le phénobarbital induit l’expression des gènes codant, entre autres, pour les CYP, responsables de son propre métabolisme oxydatif. 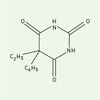 | Figure 1. Représentation schématique de la molécule de phénobarbital. |
Une autre caractéristique originale du phénobarbital est sa capacité d’induire l’expression de CYP dans toutes les espèces animales analysées, depuis la bactérie Bacillus megaterium jusqu’aux insectes, poissons, oiseaux et mammifères [3]. Un déterminant structural de la molécule de phénobarbital serait donc un déclencheur universel de ce phénomène d’induction, malgré les grandes différences existant entre les mécanismes moléculaires sous-jacents. C’est d’ailleurs en raison de la capacité du phénobarbital d’induire des gènes CYP chez Bacillus megaterium que l’on a espéré que ce processus, une fois élucidé chez la bactérie, serait transposable à l’homme. En effet, une séquence d’ADN, la boîte Barbie, présente dans la région 5’ proximale des gènes CYP de Bacillus megaterium [10] et responsable de leur activation transcriptionnelle, a été retrouvée dans les régions 5’ flanquant de nombreux gènes codant pour des CYP et pour d’autres enzymes de détoxification dans d’autres espèces, dont les rongeurs [6, 11]. Cependant, cet élément de séquence n’est pas déterminant pour l’induction des CYP par le phénobarbital chez les mammifères. Une autre région génomique plus distale, le PBRU (phenobarbital responsive unit), chez le rat, ou le PBREM (phenobarbital responsive enhancer module), chez la souris, est en effet requise [12, 13]. Cette séquence permet l’activation transcriptionnelle des gènes CYP qui possèdent un (des) site(s) de fixation pour un récepteur nucléaire orphelin, la protéine CAR (constitutive androstane receptor) impliquée dans un complexe hétérodimérique avec la molécule RXR (retinoid X receptor)(→). Un autre récepteur nucléaire, PXR (pre-gnane X receptor), encore appelé SXR (steroid X receptor), récemment identifié, permet l’induction par le phénobarbital d’une autre sousfamille de gènes CYP, là encore grâce à sa capacité d’interagir avec des séquences d’ADN spécifiques sous la forme d’un hétérodimère avec RXR (→→)[14]. Les fonctions de CAR et de PXR sont partiellement interchangeables. L’invalidation des gènes CAR et PXR démontre leur rôle central dans l’induction des gènes CYP, respectivement par le phénobarbital et par les antiglucocorticoïdes. De nombreuses revues analysant la réponse au phénobarbital ont été publiées dernièrement [6, 11, 15–18]. (→) m/s 1998, n°11, p. 1211 et 2000, n°12, p. 1413 (→→) m/s 1999, n°1, p. 113 et 2000, n°11, p. 1271 Nous nous attacherons ici à présenter une synthèse des connaissances acquises sur les mécanismes de l’induction des gènes CYP par le phénobarbital, et à soulever certaines questions importantes demeurées pour l’instant sans réponse. |
Le phénobarbital est un inducteur transcriptionnel Les premières études moléculaires ex vivo ont démontré que le phénobarbital induit une augmentation des transcrits des gènes CYP2B1, CY2B2 et CYP2C6 dans les cellules d’hépatome de rat, et CYP2H1 dans les hépatocytes de poulet [19, 20]. Cette induction est inhibée par l’α-amanitine dans les cellules d’hépatome de rat, suggérant une régulation de nature transcriptionnelle [21], comme in vivo [22]. Des expériences de transfection de plasmides recombinants ont permis de montrer que les éléments nécessaires à la réponse au phénobarbital des gènes CYP2B1, CYP2B2 et CYP2H1 sont situés vers -1,4 kb pour les gènes de rat ou de façon beaucoup plus distale (-5,9 kb) dans le cas du gène de poulet [21, 23]. Par transgenèse chez la souris, il a été montré que les séquences nécessaires à l’activation transcriptionnelle par le phénobarbital sont localisées à -0,8 kb du gène CYP2B2 de rat, et à l’intérieur d’un fragment de 19 kb contenant cette séquence, en accord avec les données obtenues par culture cellulaire [24]. L’amélioration des conditions de culture d’hépatocytes de rat et de souris a permis de démontrer que les éléments de réponse les plus importants sont localisés à la position -2,3 kb en amont du site d’initiation de la transcription des gènes CYP2B1/2 de rat et CYP2B10 de souris [12, 13]. |
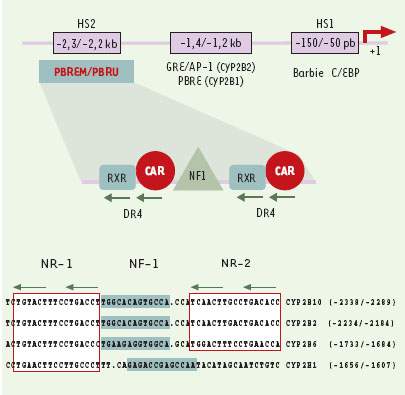
Les principaux éléments de réponse au phénobarbital sont localisés au sein d’une courte séquence de 51 paires de bases La première caractérisation de l’élément PBRE (PB responsive element) dans le promoteur du gène CYP2B10 de souris a montré que la région située entre -1,4 et -0,97 kb contient une séquence de 25 pb portant un site de fixation pour un récepteur nucléaire (NR) [13]. Un autre motif NR situé vers -2,3 kb en amont des gènes CYP2B1/2 (PBRU) et CYP2B10 (PBREM) a ensuite été identifié [11]. Les éléments PBRU et PBREM présentent 98 % d’identité (50 bases identiques sur 51). La position de ces éléments de séquence correspond à la présence d’un site d’hypersensibilité à la DNAse I (Figure 2). Des expériences de délétion et de recomposition synthétique des séquences ont montré que la région proximale a une forte activité de transcription constitutive tandis que la région distale se comporte comme un répresseur en l’absence de phénobarbital ou de ses agonistes comme le 1,4-bis[2-(3,5-dichloropyridyloxy)] benzène (TCPOBOP). La majorité de la séquence à effet négatif, importante en l’absence de TCPOBOP, est cependant comprise dans l’intervalle -0,8 kb à -1 kb [13], en accord avec les résultats obtenus par transgenèse chez la souris [24]. Récemment, un candidat répresseur susceptible de se lier à cette séquence, la protéine RBJ-Jκ, a été identifié [25]. La séquence du PBREM du gène CYP2B10 a été restreinte à un élément d’une longueur minimale de 51 pb nécessaire et suffisant pour conférer l’induction par le phénobarbital à un promoteur hétérologue. Le PBREM du gène CYP2H1 de poulet (localisé à la position -1,64 kb) permet également l’induction d’un promoteur transcriptionnel hétérologue [26], bien que des résultats partiellement contradictoires aient été rapportés [27]. Enfin, l’injection directe dans le foie de rat de plasmides recombinants porteurs du PBRU du gène CYP2B2 a confirmé que cette séquence suffisait à reproduire l’induction du gène endogène [28]. Cet ensemble d’approches expérimentales convergentes démontre que les déterminants principaux de l’induction de gènes CYP par le phénobarbital sont contenus dans les séquences PBREM/PBRU.  | Figure 2. Organisation génomique de la région 5’ des gènes CYP2B. Les distances par rapport au site d’initiation de la transcription (+1) sont indiquées en paires de bases (pb) ou en kilobases (kb). La partie centrale de la figure représente un grossissement de la région PBREM/PBRU, avec une représentation schématique de la fixation des protéines impliquées. La partie basse de la figure contient les séquences alignées contenues dans les éléments PBREM/PBRU des gènes CYP2B et CYP2H. Les sites de reconnaissance par les récepteurs nucléaires sont encadrés. Les sites de fixation de NF-1 sont en vert. HS : DNAse I hypersensitive site ; C/EBP : CAAT enhancer binding protein ; Barbie : boîte Barbie ; GRE : glucocorticoid response element ; AP-1 : activator protein 1 ; PBRE : phenobarbital responsive element ; PBREM : phenobarbital responsive enhancer module ; PBRU : phenobarbital responsive unit ; RXR : reti-noid X receptor ; CAR : constitutive androstane receptor ; NF-1 : nuclear factor 1 ; NR : nuclear receptor site ; DR : direct repetition. |
|
Le PBREM/PBRU contient des sites NR encadrant un site NF-1 Au sein du PBREM/PBRU, deux sites NR encadrent un site NF-1 [17]. Une analyse moléculaire par mutagenèse dirigée et empreinte à la DNAse I a permis de montrer que des mutations dans le site NF-1 et les sites NR abolissent l’activité enhancer du PBREM/PBRU après traitement par le TCPOBOP [29]. Les sites NR se comportent comme des répresseurs en l’absence de TCPOBOP, tandis que le site NF-1 est un site activateur [29, 30]. Récemment, il a été montré que deux éléments de séquence additionnels sont nécessaires, au moins dans les hépatocytes de rat en culture primaire, pour conférer une induction maximale. Il s’agit d’un GRE (glucocorticoid response element) immédiatement en 5’ du PBRU, et d’une répétition opposée de type ER7 légèrement plus en amont (positions -2282 à -2264 pb du gène CYP2B2) [31]. |
Le récepteur nucléaire CAR se fixe spécifiquement sur le PBREM/PBRU En recherchant les récepteurs nucléaires susceptibles de se fixer sur le PBREM, le groupe de Negishi a démontré, par des expériences de mutagenèse dirigée et de transfection dans des hépatocytes de souris, l’implication d’un motif de type DR4 (répétitions directes séparées par 4 pb) dans la réponse au phénobarbital [32]. Ces séquences sont connues pour fixer plusieurs types de récepteurs nucléaires sous la forme d’hétérodimères avec le récepteur RXR, comme par exemple le récepteur de l’hormone thyroïdienne, le récepteur LXR (liver X receptor) ou encore le récepteur de la vitamine D [33]. Des analyses de retard sur gel ont permis de montrer que RXR se fixe sur le site NR-1 dans les échantillons traités par le phénobarbital. De plus, CAR, un récepteur orphelin enrichi dans le foie, fait partie d’un complexe hétérodimérique avec RXR. L’intensité de la bande retardée augmente avec le temps de traitement par le phénobarbital. En outre, CAR subit une translocation quantitative dans le noyau sous l’effet du phénobarbital et d’agonistes [34]. L’acide okadaïque (OA), un puissant inhibiteur des sérine/thréonine phosphatases PP1 et PP2A, bloque l’étape de translocation, et abolit toute induction du gène CYP2B10 [11], suggérant ainsi que des événements de phosphorylation sont déterminants dans le mécanisme d’activation du PBREM. CAR est également capable de transactiver le PBREM du gène CYP2B6 humain ainsi que l’élément NR1 du gène CYP3A4 - un gène connu pour être induit principalement par des stéroïdes et des antibiotiques macrolides comme la rifampicine, et plus faiblement par le phénobarbital - sous l’effet du phénobarbital et de composés agonistes [15, 35]. En conséquence, CAR pourrait être un médiateur universel de l’effet du phénobarbital chez les rongeurs et chez l’homme. En outre, CAR permet la surexpression d’autres gènes en réponse au phénobarbital, dont le gène codant pour l’UDP-glucuronosyl transférase et l’énoyl-CoA hydratase [36]. Cependant, la présence éventuelle de séquences de type DR4 n’a pas été rapportée pour ces gènes, bien que des séquences de type DR1 soient présentes dans le promoteur de l’énoyl-CoA hydratase [37]. Les déterminants de séquence des gènes CYP2B ainsi que les protéines susceptibles de s’y fixer sont représentés sur la Figure 2. Bien que CAR soit l’élément le plus important dans la réponse des gènes CYP2B au phénobarbital, il faut considérer que le récepteur RXR pourrait être plus limitant encore dans la mesure où il interagit avec de nombreux autres récepteurs que CAR. Par exemple, le traitement d’hépatocytes de rat par le dérivé 9-cis de l’acide rétinoïque, un ligand du complexe RAR (retinoic acid receptor)/RXR, empêche toute induction de CYP2B2 par le phénobarbital, vraisemblablement en réduisant le taux de RXR disponible [38]. De plus, des souris porteuses d’un allèle conditionnel nul de RXR sont défectueuses dans les voies d’activation de CAR et de PXR dans le foie (voir plus loin) [39]. |
CAR est un répresseur transcriptionnel CAR est probablement complexé à des agonistes inverses de type stéroïdien (androstanes) qui sont supposés induire la dissociation de co-activateurs. Ces ligands naturels n’interfèrent pas avec l’hétérodimérisation ou la liaison à l’ADN de CAR, mais ils permettraient la transformation du complexe en une forme inactive après dissociation du co-activateur SRC-1 [40]. Cette observation pourrait donc rendre compte de l’effet de répresseur transcriptionnel de CAR lié aux agonistes inverses, la répression étant levée en présence d’inducteurs de type phénobarbital. Le gène CAR a été invalidé chez la souris C57BL/6J [41]. Les animaux de différents génotypes sont nés à la fréquence mendélienne attendue, mâles et femelles étant fertiles. La réponse au TCPOBOP du gène CYP2B10 est complètement abolie, tandis que le niveau basal d’expression est diminué, suggérant une contribution positive partielle de CAR à l’expression normale de ce gène. L’hépatomégalie et l’augmentation de synthèse d’ADN, deux réponses classiques aux composés de type phénobarbital, ne sont plus observées chez les animaux CAR−/−. Il est remarquable de n’observer aucune compensation par un autre récepteur nucléaire chez ces animaux. En particulier, le récepteur PXR, qui présente une spécificité de liaison de ligand partiellement redondante avec celle de CAR, ainsi qu’une capacité de transactivation du PBREM (voir plus loin) ne permet pas l’induction. Ces résultats démontrent une forte spécificité de la réponse au phénobarbital via la mobilisation de CAR comme un élément central. |
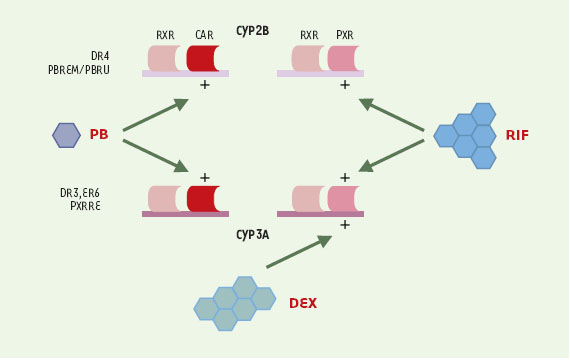
Les récepteurs nucléaires sont des capteurs des signaux engendrés par les xénobiotiques Outre CAR, un autre membre de la famille des récepteurs orphelins a été découvert, le récepteur PXR qui se lie à des sites DR3 (répétitions directes séparées par 3 pb) et ER6 (répétitions en orientation inverse et vers l’extérieur, séparées par 6 pb) et transactive le gène CYP3A en réponse à ses inducteurs spécifiques (stéroïdes, antibiotiques) [14]. Comme CAR, PXR forme un hétérodimère avec RXR, et CAR et PXR peuvent partiellement se substituer pour la liaison à l’ADN et pour la réponse au phénobarbital [35, 42]. Cependant, bien que PXR puisse être activé par la dexaméthasone, un inducteur de certains gènes CYP2B dans les hépatocytes, CAR n’est pas activé par l’hormone. De plus, ni la dexaméthasone ni PXR n’activent efficacement le PBREM [18]. En outre, chez des souris dont le gène codant pour le récepteur des hormones glucocorticoïdes (GR) a été invalidé, l’induction des gènes CYP2B par les stéroïdes n’est plus observée, tandis que celle des gènes CYP3A est maintenue [43]. Cependant, dans d’autres études, il a été montré que l’hétérodimère PXR/RXR peut se fixer sur le PBREM/PBRU de rongeur et humain avec une spécificité de liaison comparable à celle de CAR/RXR [44], cette fixation conduisant à une stimulation de la transcription d’un gène rapporteur. Le PXR activé par la rifampicine est capable d’augmenter l’activité du promoteur CYP2B dans des cellules CV-1 et dans des hépatocytes de rat en culture primaire. Des expériences en miroir montrent que CAR/RXR peut se fixer sur les sites de reconnaissance de PXR/RXR présents dans le promoteur des CYP3A, et induit l’activité du promoteur CYP3A. Cette induction est bloquée par l’androsténol, mais stimulée par le TCPOBOP, comme il est attendu des caractéristiques fonctionnelles de CAR. De même, le gène CYP3A est induit par le phénobarbital mais pas par la dexaméthasone dans le foie de souris PXR−/−, suggérant l’intervention possible de CAR ou du GR. En outre, chez des souris exprimant un transgène PXR dans le foie, on observe une expression accrue du gène CYP2B10, et une stimulation par un ligand spécifique de PXR. Enfin, l’induction de CYP2B10 par le phénobarbital est plus forte dans le foie de souris PXR −/−, ce qui pourrait suggérer une interférence négative entre les deux voies d’activation. Des expériences complémentaires sont requises pour évaluer plus complètement le degré d’interchangeabilité de CAR et de PXR. L’existence éventuelle de différences dans les niveaux d’expression et/ou de disponibilité des différents récepteurs au cours du développement, selon les tissus, et leur besoin en co-activateurs ou co-répresseurs, demeure une question centrale non encore élucidée. Un schéma hypothétique des relations entre CAR et PXR est présenté sur la Figure 3. 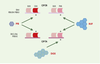 | Figure 3. Schéma hypothétique des relations entre CAR et PXR influençant l’expression des gènes CYP2B et CYP3A. Les hétérodimères CAR/RXR et PXR/RXR seraient tous deux capables d’interagir avec les séquences DR4 (CYP2B PBREM/PBRU) et DR3, ER6 (CYP3A, PXRRE). La spécificité serait modulée par les inducteurs phénobarbital (PB), rifampicine (RIF) et dexaméthasone (DEX). Les éléments DR3 et ER6 présents dans les promoteurs des gènes CYP3A ont les séquences suivantes : |
En tant qu’acteurs dans une réponse cellulaire intégrée, les récepteurs nucléaires et les CYP sont engagés dans des mécanismes de cross-talk. En effet, plusieurs CYP dégradent ou produisent des ligands de récepteurs nucléaires directement responsables de l’induction, ou de la répression de gènes CYP de familles distinctes [17]. Par exemple, les CYP1A2 et les CYP2C19 produisent des dérivés œstrogéniques à partir du méthoxychlor, un agoniste de CAR, en dépit du fait que ces deux CYP ne sont pas stimulés par CAR. |
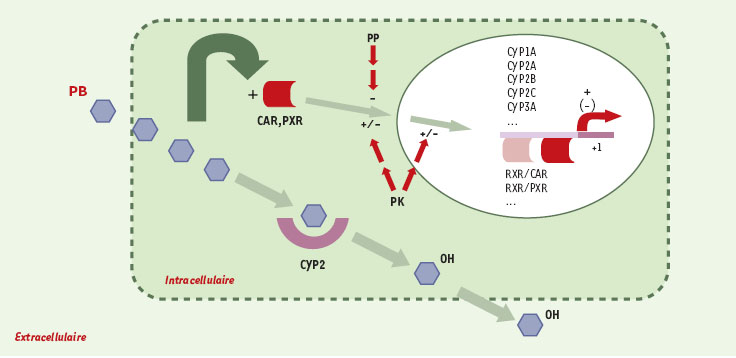
Plusieurs voies de transduction sont mobilisées sous l’effet du phénobarbital La 2-aminopurine, un inhibiteur général des sérine/thréonine protéine kinases bloque l’induction des CYP2B1/2 dans les hépatocytes de rat en culture primaire [45]. La génistéine, un inhibiteur des tyrosine kinases empêche également toute induction, suggérant un rôle important d’isoformes des protéine kinases A (PKA) ou C (PKC) dans cette induction. Cependant, d’autres études n’ont pas mis en évidence d’effet de la génistéine sur l’induction des CYP2B ou 2H par le phénobarbital [46, 47]. L’induction des CYP2B et des CYP3A par le phénobarbital est pratiquement abolie par des traitements qui augmentent le taux d’AMPc dans les hépatocytes de rat et de souris [48, 49]. Des résultats similaires sont observés en réponse au 8-bromo-AMPc, dibutyryl AMPc et 8-CPT-AMPc [48]. La PKA exerce donc un effet négatif sur l’induction par le phénobarbital. De même, l’acide okadaïque, un puissant inhibiteur des sérine/ thréonine phosphatases PP1 et PP2A bloque également toute induction des gènes CYP2B1/2 et CYP2B10 [34, 50]. Cet effet suppresseur potentialise l’effet négatif du dibutyril AMPc [34]. Comme cela a déjà été discuté, la translocation de CAR dans le noyau est bloquée par l’acide okadaïque, ce qui suggère que CAR pourrait en être la cible principale. Cependant, l’existence d’un lien entre le niveau de phosphorylation de CAR (ou PXR) et son activité, en particulier en réponse à l’activation des protéine kinases, n’a pas été analysée. Par ailleurs, l’inhibition de la calmoduline kinase dépendante du calcium, par le KN62 ou par le BAPTA-AM, un chélateur du calcium intracellulaire, inhibe toute induction des gènes CYP2B10 et CYP3A dans les hépatocytes de souris en culture primaire, indiquant un rôle positif du calcium et de la protéine kinase II [49]. En revanche, les inhibiteurs de la protéine kinase C (H-7), de la PI-3 kinase (LY-294002) et de la MAP-kinase (PD-98059) sont sans effet sur l’induction par le phénobarbital du gène CYP2B10 (Figure 4) [49]. 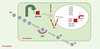 | Figure 4. Schéma récapitulatif et hypothétique de la voie de transduction du signal induite par le phénobarbital. Le phénobarbital entrerait passivement dans la cellule et activerait les récepteurs CAR et PXR, selon un (des) mécanisme(s) sensible(s) à des activités de type kinase et phosphatase. La fixation des complexes hétérodimériques CAR/RXR et PXR/RXR sur leurs séquences cibles situées en amont des gènes CYP2 et CYP3A induirait l’activation transcriptionnelle de ces gènes. On notera également que le phénobarbital est un substrat de gènes CYP dont il induit l’expression (CYP2B), ce qui pourrait concourir à augmenter son métabolisme. Les dérivés hydroxylés du phénobarbital quitteraient la cellule par diffusion. PP : protéine phosphatase ; PK : protéine kinase. |
|
Contrôle de l’expression des gènes codantpar le phénobarbital : des questions encore sans réponse Les caractéristiques fonctionnelles de CAR et de PXR demeurent partiellement inconnues. En particulier, on ne connaît ni l’éventail des gènes stimulés ou inhibés par ces récepteurs sous l’effet du phénobarbital, ni les mécanismes de régulation d’expression de CAR et de PXR dans le foie, et potentiellement dans d’autres tissus comme le cerveau où des gènes CYP sont inductibles par le barbiturique [51]. Un élément de réponse a été apporté récemment par le groupe de M.J. Villarem qui a démontré que les hormones glucocorticoïdes stimulent le taux d’expression des transcrits de CAR dans les hépatocytes humains en culture [52]. Ce traitement augmente également la translocation de CAR dans le noyau, ainsi que la réponse au phénobarbital des gènes CYP2B6 et CYP3A4, un effet bloqué par le RU486, suggérant une intervention directe du récepteur des glucocorticoïdes. Il apparaît donc qu’un lien fonctionnel entre les voies contrôlées par CAR et PXR soit établi par le récepteur GR. En revanche, la capacité éventuelle du phénobarbital d’induire l’expression de CAR ou de PXR n’a pas été rapportée. Par ailleurs, le mécanisme d’induction par le phénobarbital n’est pas universel. En effet, la séquence du PBREM du gène CYP2B9 n’est pas fonctionnelle en raison de nombreuses mutations par rapport à celle du CYP2B10 [53]. Cependant, l’expression du CYP2B9 peut être induite dans certaines lignées de souris consanguines [54], tandis que la séquence de son PBREM est identique dans les lignées inductibles et non inductibles. Se pose donc la question de la présence éventuelle d’un autre site de fixation de CAR et/ou de PXR dans le gène CYP2B9, ou celle de la mise en place d’un mécanisme d’induction alternatif. Enfin, la composante génétique du mécanisme d’induction par le phénobarbital reste peu explorée. L’induction du gène codant pour l’aldéhyde déshydrogénase de type II (ALD-2) par le phénobarbital chez le rat est sous le contrôle d’un locus génétique majeur à effet semi-dominant [55]. Chez les animaux homozygotes R/R, l’induction du gène ALD-2 est très diminuée par rapport aux animaux de génotype r/r, et, observation remarquable, l’expression du gène CYP2B2 est inhibée par le phénobarbital tandis que celle du gène CYP2B1 est induite, comme chez les animaux de génotype r/r. Ces observations sont en accord avec l’existence supposée d’un gène modificateur, et ne sont pas dues à l’existence de mutations dans la séquence du PBRU. Des résultats très comparables ont été obtenus chez la souris pour les gènes orthologues CYP2B9 et ALD-2 [56]. L’analyse génétique a permis d’identifier une région porteuse d’un locus régulateur potentiel sur le chromosome 7, dans une région éloignée des gènes CYP2B, et indépendante génétiquement du gène ALD-2 porté par le chromosome 19 [57]. Le (ou les) gène(s) porté(s) par ce locus restent à identifier. |
Notre compréhension des mécanismes qui opèrent lors de la réponse des gènes CYP2B et CYP3A aux inducteurs de type phénobarbital s’est améliorée ces dernières années. Le rôle central joué par les récepteurs nucléaires orphelins CAR et PXR dans cette induction s’est révélé bien après que les approches pharmacologiques aient permis de formuler l’hypothèse de leur existence. L’avenir devra cependant s’attacher à répondre aux nombreuses questions encore en suspens, en particulier en ce qui concerne la population humaine. Les paramètres génétiques qui établissent ou concourent à la variabilité interindividuelle de réponse aux médicaments devront être déterminés. Dès lors que l’inflammation et les maladies chroniques sont souvent le terreau sur lequel les médicaments doivent exercer leurs effets curatifs, il faudrait disposer de systèmes prédictifs aisément accessibles au cas par cas. Cela permettrait d’anticiper la réponse attendue, surtout en cas d’interactions médicamenteuses qui résultent souvent de phénomènes d’induction ou d’inhibition enzymatique. |
1. Conney AH. Induction of microsomal enzymes by foreign chemicals and carcinogenesis by polycyclic aromatic hydrocarbons: G.H.A. Clowes Memorial Lecture. Cancer Res 1982; 42 : 4875–917. 2. Okey AB. Enzyme induction in the cytochrome P-450 system. Pharmacol Ther 1990; 45 : 241–98. 3. Waxman DJ, Azaroff L. Phenobarbital induction of cytochrome P-450 gene expression. Biochem J 1992; 281 : 577–92. 4. Frueh FW, Zanger UM, Meyer UA. Extent and character of phenobarbital-mediated changes in gene expression in the liver. Mol Pharmacol 1997; 51 : 363–9. 5. Whysner J, Ross PM, Williams GM. Phenobarbital mechanistic data and risk assessment: enzyme induction, enhanced cell proliferation, and tumor promotion. Pharmacol Ther 1996; 71 : 153–91. 6. Kemper B. Regulation of cytochrome P450 gene transcription by phenobarbital. Prog Nucleic Acids Res Mol Biol 1998; 61 : 23–64. 7. Gonzalez FJ. The molecular biology of cytochrome P450s. Pharmacol Rev 1988; 40 : 243–88. 8. Pitot HC, Dragan YP, Teeguarden J, Hsia S, Campbell H.Quantitation of multistage carcinogenesis in rat liver. Toxicol Pathol 1996; 24 : 119–28. 9. Lee GH. Paradoxical effects of phenobarbital on mouse hepatocarcinogenesis. Toxicol Pathol 2000; 28 : 215–25. 10. He JS, Fulco AJ. Abarbiturate-regulated protein binding to a common sequence in the cytochrome P450 genes of rodents and bacteria. J Biol Chem 1991; 266 : 7864–9. 11. Honkakoski P, Negishi M. Regulatory DNA elements of phenobarbitalresponsive cytochrome P450 CYP2B genes. J Biochem Mol Toxicol 1998; 12 : 3–9. 12. Trottier E, Belzil A, Stoltz C, Anderson A. Localization of a phenobarbital-responsive element (PBRE) in the 5’-flanking region of the rat CYP2B2 gene. Gene 1995; 158 : 263–8. 13. Honkakoski P, Moore R, Gynther J, Negishi M. Characterization of phenobarbital-inducible mouse Cyp2b10 gene transcription in primary hepatocytes. J Biol Chem 1996; 271 : 9746–53. 14. Moore JT, Kliewer SA. Use of the nuclear receptor PXR to predict drug interactions. Toxicology 2000; 153 : 1–10. 15. Dogra SC, Whitelaw ML, May BK. Transcriptional activation of cytochrome P450 genes by different classes of chemical inducers. Clin Exp Pharmacol Physiol 1998; 25: 1–9. 16. Waxman DJ. P450 gene induction by structurally diverse xenochemicals: central role of nuclear receptors CAR, PXR, and PPAR. Arch Biochem Biophys 1999; 369 : 11–23. 17. Honkakoski P, Negishi M. Regulation of cytochrome P450 (CYP) genes by nuclear receptors. Biochem J 2000; 347 : 321–37. 18. Zelko I, Negishi M. Phenobarbital-elicited activation of nuclear receptor CAR in induction of cytochrome P450 genes. Biochem Biophys Res Commun 2000; 277 : 1–6. 19. Corcos L, Weiss MC. Phenobarbital, dexamethasone and benzanthracene induce several cytochrome P450 mRNAs in rat hepatoma cells. FEBS Lett 1988; 233 : 37–40. 20. Hansen AJ, May BK, Eferlink LA. Sequence of a chicken phenobarbital-inducible cytochrome P450 cDNA: regulation of two mRNAs transcribed from different genes. DNA 1989; 8 : 179–91. 21. Shaw PM, Adesnik M, Weiss MC, Corcos L. The phenobarbital-induced transcriptional activation of cytochrome P-450 genes is blocked by the glucocorticoid-progesterone antagonist RU486. Mol Pharmacol 1993; 44 : 775–83. 22. Hardwick J P, Gonzalez FJ, Kasper CB. Transcriptional regulation of rat liver epoxide hydratase, NADPH-cytochrome P-450 oxidoreductase, and cytochrome P-450b genes by phenobarbital. J Biol Chem 1983; 258 : 8081–5. 23. Hahn CN, Hansen AJ, May BK. Transcriptional regulation of the chicken CYP2H1 gene. J Biol Chem 1991; 266 : 17031–9. 24. Ramsden R, Sommer KM, Omiecinski CJ. Phenobarbital induction and tissue-specific expression of the rat CYP2B2 gene in transgenic mice. J Biol Chem 1993; 268 : 21722–6. 25. Lee SH, Wang X, DeJong J. Functional interactions between an atypical NF-kappaB site from the rat CYP2B1 promoter and the transcriptional repressor RBP-Jkappa/CBF1. Nucleic Acids Res 2000; 15 : 2091–8. 26. Handschin C, Podvinec M, Meyer UA. CXR, a chicken xenobiotic-sensing orphan nuclear receptor, is related to both mammalian pregnane X receptor (PXR) and constitutive androstane receptor (CAR). Proc Natl Acad Sci USA 2000; 97 : 10769–74. 27. Dogra SC, Benjamin P, Davidson P, May BK. Analysis of a phenobarbital-responsive enhancer sequence located in the 5’ flanking region of the chicken CYP2H1 gene: identification and characterization of functional protein-binding sites. Mol Pharmacol 1999; 55 : 14–22. 28. Park Y, Li H, Kemper B. Phenobarbital induction mediated by a distal CYP2B2 sequence in rat transiently transfected in situ. J Biol Chem 1996; 271 : 23725–8. 29. Honkakoski P, Moore R, Washburn K, NegishiM. Activation by diverse xenochemicals of the 51- bp phenobarbitalresponsive enhancer module of the Cyp2b10 gene. Mol Pharmacol 1998; 53 : 597–601. 30. Honkakoski P, Negishi M. Characterization of a phenobarbital-responsive enhancer module in mouse P450 Cyp2b10 gene. J Biol Chem 1997; 272 : 14943–9. 31. Paquet Y, Trottier E, Beaudet MJ, Anderson A. Mutational analysis of the CYP2B2 phenobarbital response unit and inhibitory effect of constitutive androstane receptor on phenobarbital responsiveness. J Biol Chem 2000; 275 : 38427–36. 32. Honkakoski P, Zelko I, Sueoshi T, Negishi M. The nuclear orphan receptor CAR-retinoid X receptor heterodimer activates the phenobarbitalresponsive enhancer module of the CYP2B gene. Mol Cell Biol 1998; 18 : 5652–8. 33. Quack M, Carlberg C. Ligand-triggered stabilization of vitamin D receptor/retinoid X receptor heterodimer conformations on DR4-type response elements. J Mol Biol 2000; 296 : 743–56. 34. Kawamoto T, Sueyoshi T, Zelko I, Moore R, Washburn K, Negishi M. Phenobarbital-responsive nuclear translocation of the receptor CAR in induction of the CYP2B gene. Mol Cell Biol 1999; 19: 6318–22. 35. Sueyoshi T, Kawamoto T, ZelkoI I, Honkakoski P, Negishi M. The repressed nuclear receptor CAR responds to phenobarbital in activating the human CYP2B gene. J Biol Chem 1999; 274 : 6043–6. 36. Kassam A, Winrow CJ, Fernandez-Rachubinski F, Capone JP, Rachubinski RA. The peroxisome proliferator response element of the gene encoding the peroxisomal betaoxidation enzyme enoyl-CoA hydratase/3-hydroxyacyl-CoA dehydrogenase is a target for constitutive androstane receptorbeta/9-cisretinoic acid receptormediated transactivation. J Biol Chem 2000; 275 : 4345–50. 37. Zhang B, Marcus SL, Sajjadi FG, et al. Identification of a peroxisome proliferatorresponsive element upstream of the gene encoding rat peroxisomal enoyl-CoA hydratase/3-hydroxyacyl-CoA dehydrogenase. Proc Natl Acad Sci USA 1992 : 7541–7545. 38. Yamada H, Yamaguchi T, Oguri K. Suppression of the expression of the CYP2B1/2 gene by retinoic acids. Biochem Biophys Res Commun 2000; 277 : 66–71. 39. Wan YJ, Yu-Jui Y, Dahsing A, et al. Hepatocytespecific mutation establishes retinoid X receptor a as a heterodimeric integrator of multiple physiological processes in the liver. Mol Cell Biol 2000; 20 : 4436–44. 40. Forman BM, Tzameli I, Choi HS, et al. Androstane metabolites bind to and deactivate the nuclear receptor CAR-beta. Nature 1998; 395 : 612–5. 41. Wei P, Zhang J, Egan-Hafley M, Liang S, Moore DD. The nuclear receptor CAR mediates specific xenobiotic induction of drug metabolism. Nature 2000; 407 : 920–3. 42. Moore LB, Park DJ, Jones SA, et al. Orphan nuclear receptors constitutive androstane receptor and pregnane X receptor share xenobiotic and steroid ligands. J Biol Chem 2000; 275 : 15122–7. 43. Schuetz EG, Schmid W, Schutz G, et al. The glucocorticoid receptor is essential for induction of cytochrome P-4502B by steroids but not for drug or steroid induction of CYP3A or P-450 reductase in mouse liver. Drug Metab Dispos 2000; 28 : 268–78. 44. Xie W, Barwick JL, Simon CM, et al. Reciprocal activation of xenobiotic response genes by nuclear receptors SXR/PXR and CAR. Genes Dev 2000; 14 : 3014–23. 45. Nirodi CS, Sultana S, Ram N, Prabhu L, Padmanaban G. Involvement of synthesis and phosphorylation of nuclear protein factors that bind to the positive cis-acting element in the transcriptional activation of the CYP2B1/B2 gene by phenobarbitone in vivo. Arch Biochem Biophys 1996; 331 : 79–86. 46. Kawamura A, Yoshida Y, Kimura N, Oda H, Kakinuma A. Phosphorylation/ dephosphorylation steps are crucial for the induction of CYP2B1 and CYP2B2 gene expression by phenobarbital. Biochem Biophys Res Commun 1999; 264 : 530–6. 47. Dogra SC, May BK. Phenobarbital-induced activation of CYP2H1 and 5-aminolevulinate synthase genes in chick embryo hepatocytes is blocked by an inhibitor of protein phosphorylation. Arch Biochem Biophys 1996; 327 : 271–8. 48. Sidhu JS, Omiecinski CJ. cAMP-associated inhibition of phenobarbital-inducible cytochrome P450 gene expression in primary rat hepatocyte cultures. J Biol Chem 1995; 270 : 12762–73. 49. Marc N, Galisteo M, Lagadic-Gossmann D, et al. Regulation of phenobarbital induction of the cytochrome P450 2b9/10 genes in primary mouse hepatocyte culture. Involvement of calciumand cAMP-dependent pathways. Eur J Biochem 2000; 267 : 963–70. 50. Sidhu JS, Omiecinski CJ. An okadaic acid-sensitive pathway involved in the phenobarbital-mediated induction of CYP2B gene expression in primary rat hepatocyte cultures. J Pharmacol Exp Ther 1997; 282 : 1122–9. 51. Geng J, Strobel HW. Expression, induction and regulation of the cytochrome P450 monooxygenase system in the rat glioma C6 cell line. Brain Res 1998; 784 : 276–83. 52. Pascussi JM, Gerbal-Chaloin S, Fabre JM, Maurel P, Vilarem MJ. Dexamethasone enhances constitutive androstane receptor expression in human hepatocytes: consequences on cytochrome P450 gene regulation. Mol Pharmacol 2000; 58 : 1441–1450. 53. Damon M, Fautrel A, Guillouzo A, Corcos L. Genetic analysis of the phenobarbital regulation of the cytochrome P-450 2b-9 and aldehyde dehydrogenase type 2 mRNAs in mouse liver. Biochem J 1996; 317 : 481–6. 54. Dunn TJ, Koleske AJ, Lindahl R, Pitot HC. Phenobarbitalinducible aldehyde dehydrogenase in the rat. cDNA sequence and regulation of the mRNA by phenobarbital in responsive rats. J Biol Chem 1989; 264 : 13057–65. 55. MarcN, Fautrel A, Damon M, Guillouzo A, Corcos L. Phenobarbital induction of aldehyde dehydrogenase type 2 mRNA in mouse liver: a candidate region on chromosome 7 for a putative regulatory gene. Biochem Genet 2000; 38 : 288–98. |